近日,实验室蒋宇扬教授与新加坡国立大学陈宇综教授合作,开发了一款新颖的化合物计算机特征表示的方法MolMap,在药物相关性质预测方面表现出色,并基于此开发了开箱即用(Out-of-the-Box)、操作方便的化合物深度学习模型MolMapNet, 方便非AI专业研究人员的使用。

MolMap分子表征和开箱即用模型示意图
药物研发是一个漫长而复杂的过程, 而深度学习方法已被推广应用到提高药物发现的速度和能力方面。但成功的药物深度学习主要依赖于对化合物的表征的充分解析,最近兴起了使用图卷积神经网络(GCN, 基于图形的分子表示从头学习)来预测药物性质,在理化性质,活性,毒理,药代预测等方面,取得了最佳的能力。然而,人类长期积累起来的药物化学专家知识和卷积神经网络(CNNs)的优异学习能力在药物学习方面的综合潜力尚待充分挖掘。化合物的分子描述符和分子指纹来源于人类专家知识,用于全面描述分子的结构、理化、拓扑和子结构特征,这些都是学习药物特性的重要先验知识。但如何将其转化为更合适CNN深度学习的表征是一个有待解决的问题。
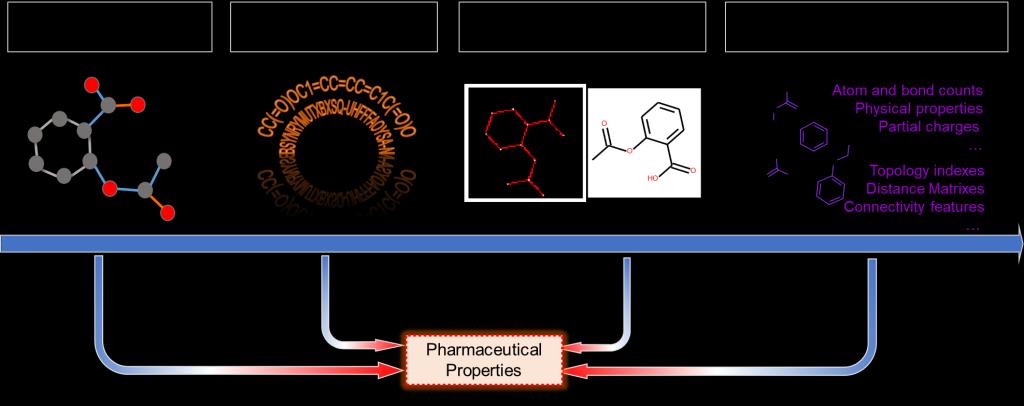
分子深度学习中用于药物性质预测的代表性的四种分子表征形式
对此,研究团队首次尝试对超过850万化合物的1,456分子描述符和16,204分子指纹进行基于流行学习嵌入(manifold embedding)的解析,建立了一套全新的的化合物特征生成方法,基于此开发了一个新的化合物深度学习工具--开箱即用MolMap,旨在将药物研究者从复杂的编程和软件优化工作中解放出来。
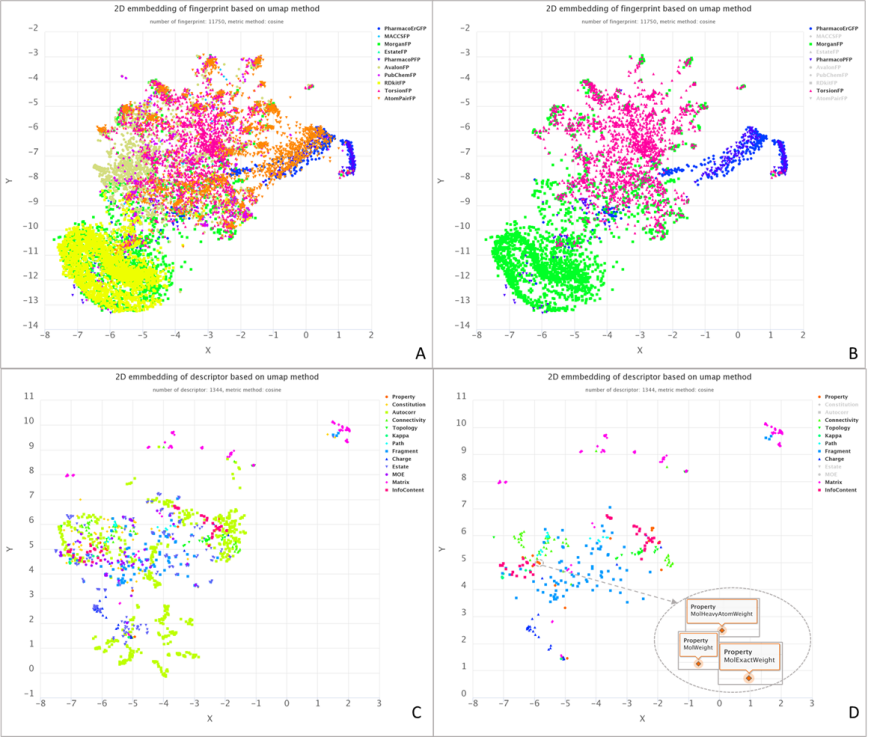
分子指纹和分子描述符的二维嵌入, A&B:分子指纹, C&D:分子描述符,每种颜色代表一种类型的描述符或分子指纹
研究团队首先将超过1.1亿分子分为100类,采用分层采样技术,根据分子的特定指纹,从100个类别中提取超过850万个采样分子。对这850万个分子计算13类结构描述符、物理化学描述符和拓扑描述符(共1,456个)和12类分子指纹(共16,204个)。基于分子对之间的距离,应用UMAP算法将无序排列的分子描述符和分子指纹投影到二维空间形成关联有序的2D排列图,再使用J-V算法分配到一个2D规则网格中,形成可输入CNN深度学习的标准2D特征图,流程图如下所示。

MolMap分子表征生成流程框架
此外,生成的2D特征图使用多通道网络可增强多帧或多类数据的深度学习性能,在MolMap中,分子描述符特征图有13个类别,可分成13个通道,分子指纹特征图聚类为3类,可分成3个通道,分类特征图如上图所示。MolMapNet包括分子描述符单路径模型、分子指纹单路径模型和分子描述符、分子指纹双路径模型,参数的最大数目不超过83万。
研究人员基于16个基准数据集上对MolMap的性能进行评价和度量,与已发布的最先进的基于图形的GCN/GAT模型在相同数据集和数据分割(训练、验证、测试)集上的性能进行比较。在12个药物相关数据集中,有9个MolMapNet表现优于GCN/GAT模型,显示了MolMaNet的优异能力。在3个理化数据集上,MolMapNet表现不如GCN/GAT模型,部分原因是MolMapNet从分子描述符中学习,其中一些是计算得到而不是实验得到的理化性质(如ClogP)。此外,作者进一步做了MolMapNet的可解释性的研究, 在β分泌酶-1(BACE1)高活性抑制剂的预测上,发现模型学到了有利于高活性的分子指纹和药效团,学习到的特征非常有利于识别高效力抑制剂,为指导药物化学家合成高效力化合物提供更好的思路:
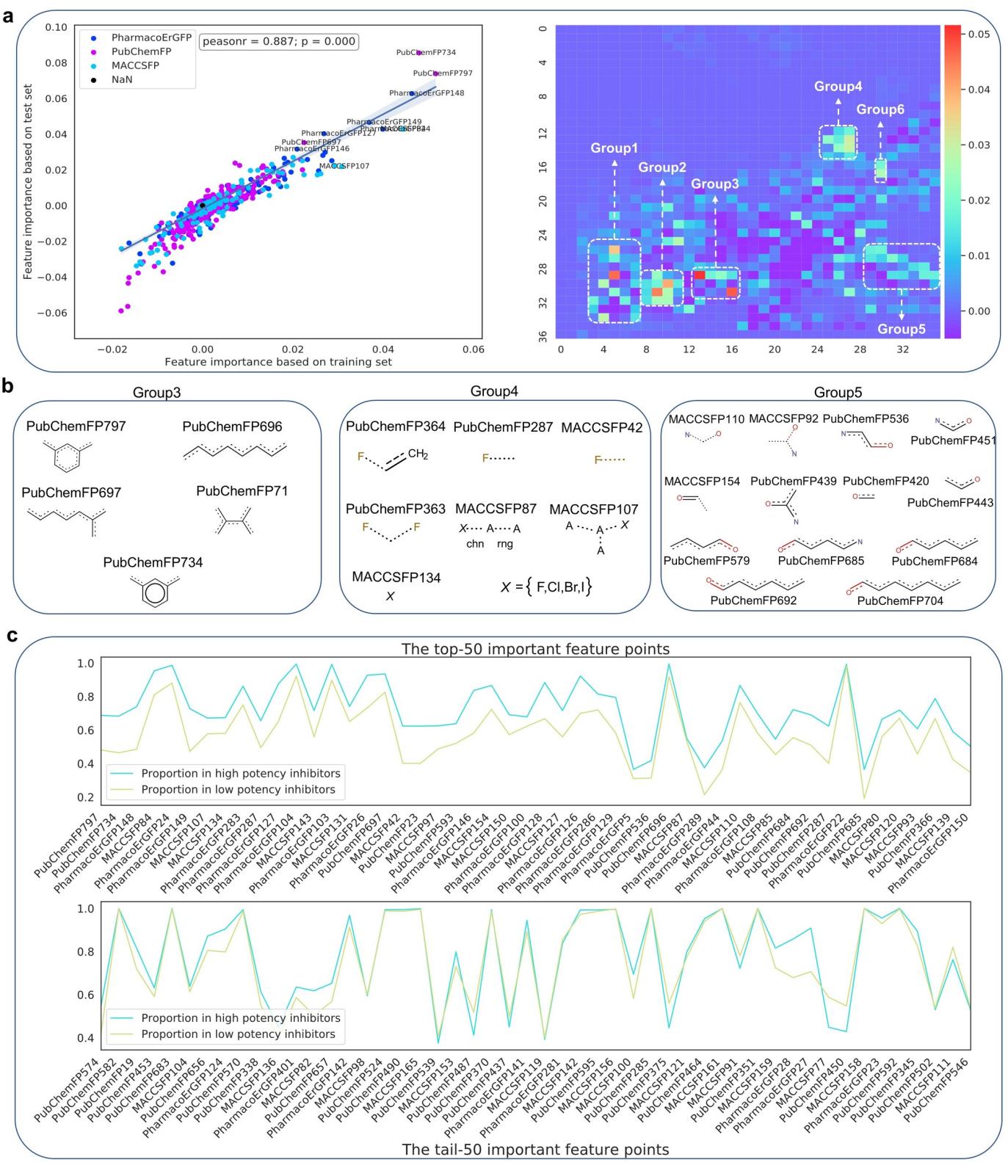
MolMapNet从BACE1数据里学到的重要的分子指纹和药效团,可用于对活性未知的小分子进行识别
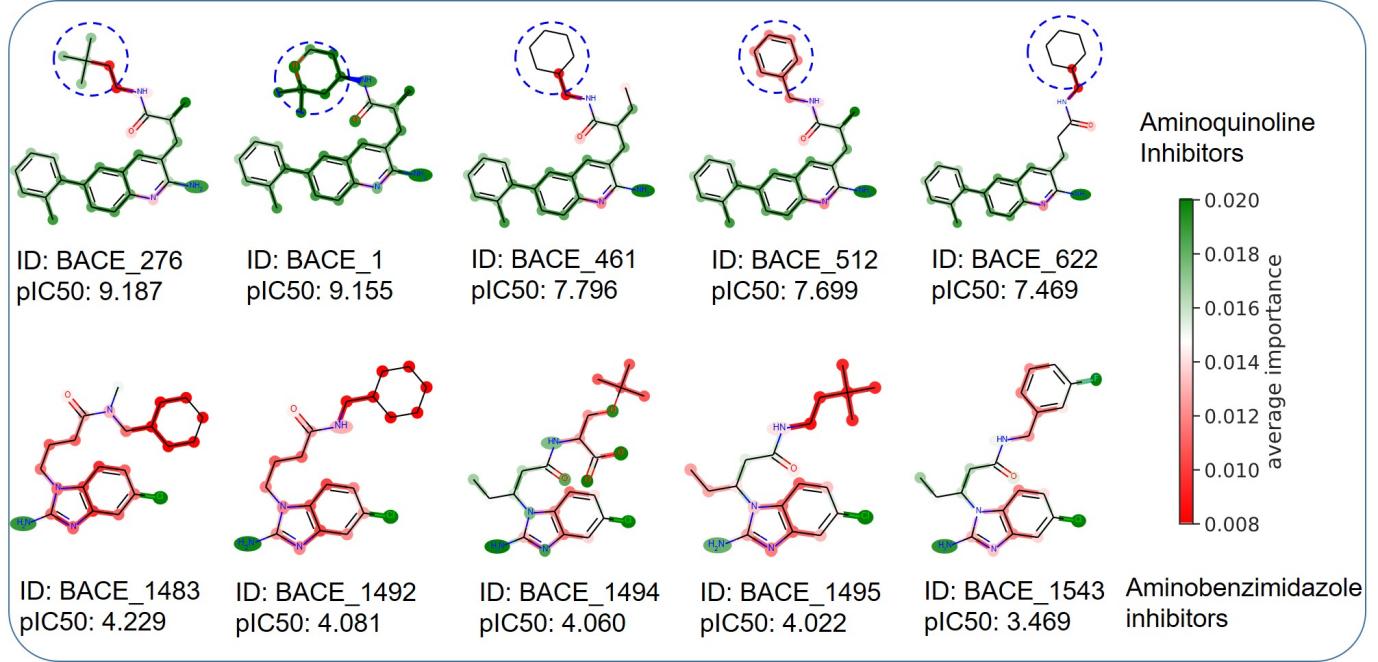
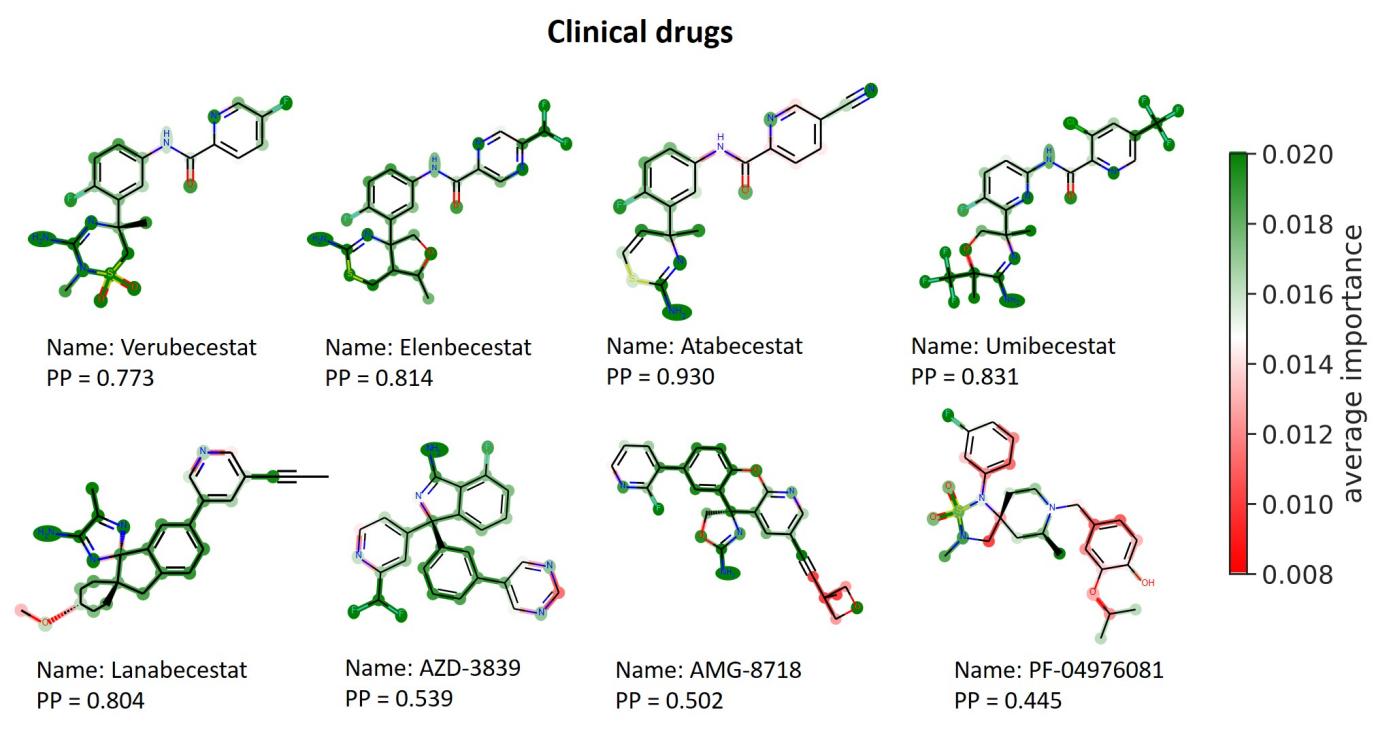
基于重要的分子指纹和药效团, MolMap 识别出 实验室合成的小分子和临床药物的活性部位结构, 绿色代表有利于活性结构,红色代表不利于高活性
据研究团队介绍,这项工作最大的亮点在于将一维无序排列数据转二维有序排列、空间相关的图像类似的数据便于高效率的AI学习,于此同时利用了两类分子性质(分子指纹和分子描述符),便于AI从两个侧面更全面的学习分子性质。同时是在药物领域建立开箱即用AI学习工具,便于小型药企及科研团队在没有AI专业知识的情况下,利用自己的内部数据建立AI药物研究工具。
2021年3月1日,本项研究成果以 “Out-of-the-box deep learning prediction of pharmaceutical properties by broadly learned knowledge-based molecular representations” 为题在线发表于 《自然-机器智能》(Nature Machine Intelligence)。本研究获得了国家重点研发计划、深圳市科技创新委基金、广东省科技基金以及新加坡学术基金的资助。
文章链接:https://www.nature.com/articles/s42256-021-00301-6